Segmentation d'image en imagerie médicale
Par Loïc Muhirwa, Statistique Canada
Introduction
Étant donné que de nombreuses applications nécessitent l'identification des substructures des images numériques, la segmentation est une procédure fondamentale de prétraitement. La segmentation binaire entre l'avant-plan et l'arrière-plan d'une image, d'application très large, est un exemple canonique de segmentation d'image. En imagerie médicale, il peut être nécessaire de segmenter des images de résonance magnétique (RM) ou de tomodensitométrie (TDM) d'un organe en structures anatomiques distinctes ou de segmenter différents types de tissus. Dans le domaine de la neuroimagerie particulièrement, il est possible de segmenter le cerveau humain selon le tissu principal (matière blanche et grise) ou selon l'état de santé d'un tissu, c'est-à-dire sain ou avec lésion.
Pour mettre en forme ces idées, il nous faut une représentation mathématique d'une image. Il existe plusieurs manières de représenter les images, selon les applications; certaines sont plus pratiques que d'autres. Comme le décrit l'article, en raison de la multiplicité des méthodes de segmentation d'image, il est difficile de trouver une représentation mathématique unique d'une image. Malgré cela, nous adopterons une représentation primaire, de laquelle nous pourrons nous écarter aux fins de simplification de la notation ou pour régler les cas dans lesquels une image est un objet discret, par opposition à un objet continu. Formellement, une image peut être représentée comme une table de pixels d'un domaine d'image à un domaine d'intensité, comme suit :
où le domaine d'image est un espace compact et un sous-ensemble simplement connexe de pour des images en 2D ou 3D, également appelé volumes (voir : la définition de simplement connexe – le contenu de cette page est en anglais). Par ailleurs, sans perte de généralité, l'article étudie seulement les images aux valeurs d'intensité unidimensionnelles, telles que les images en niveaux de gris. Il s'agit d'une définition implicite étant donné que l'image est mise en correspondance avec . Dans cette représentation d'image, nous pouvons définir conceptuellement une segmentation Z de l'image I comme étant la carte suivante :
où est le nombre de segments d'image distincts.
Figure 1 : Segmentation d'une coupe axiale du cerveau. Le rouge, le vert et le bleu correspondent respectivement à la matière blanche, à la matière grise et au liquide céphalorachidien. (Source de l'image : A review of medical image segmentation: methods and available software.Footnote 21)
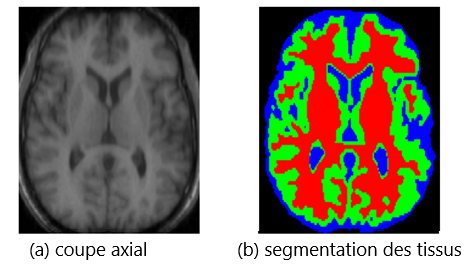
Description - Figure 1: Segmentation d'une coupe axiale du cerveau. Le rouge, le vert et le bleu correspondent respectivement à la matière blanche, à la matière grise et au liquide céphalorachidien. (Source de l'image : A review of medical image segmentation: methods and available software.
Imagerie cérébrale d'une coupe axiale à gauche en noir et blanc, et segmentation des tissus représentée par les couleurs rouge, vert et bleu à droite. Les couleurs de l'image de droite correspondent respectivement à la matière blanche, à la matière grise et au liquide céphalorachidien, de l'image de gauche.
En gros, la segmentation des images numériques peut être classée en deux catégories : la segmentation manuelle et la segmentation automatique. Dans la segmentation manuelle, un être humain annote manuellement les différents segments dans une image numérique, tandis que dans la segmentation automatique, un algorithme automatisé segmente l'image. La segmentation manuelle présente plusieurs difficultés, notamment en matière de coût, de temps et d'uniformité. Premièrement, dans de nombreuses applications, la personne qui annote les images doit être spécialiste du domaine, ce qui rend la segmentation manuelle difficile et coûteuse sur le plan opérationnel.
Si l'on prend l'exemple de la neuroimagerie médicale, certains des types de segmentation les plus courants consistent en une segmentation selon le type de tissu, qui classe le tissu cérébral en trois grands types : matière blanche, matière grise et liquide céphalorachidien (voir la figure 1). L'annotation manuelle du volume d'images neurologiques d'un seul patient nécessite beaucoup de temps et des connaissances de radiologue. Deuxièmement, le traitement d'image peut nécessiter l'annotation de centaines d'images, ce qui est ingérable en cas de traitement manuel. Troisièmement, les segmentations manuelles varient considérablement d'une personne à l'autre, y compris quand elles sont réalisées par des spécialistesFootnote 1. Certains tomodensitomètres peuvent produire des images facilement segmentables par un procédé automatique. À titre d'exemple, comme une image neurologique de tomodensitométrie a des intensités ayant une correspondance physique fixe, la segmentation automatisée ne pose pas de difficulté. Pour ces raisons et bien d'autres, il faut privilégier la segmentation automatique.
Dans les sections suivantes, nous classerons les méthodes de segmentation automatique en deux catégories : les méthodes fondées sur un modèle génératif et les méthodes d'apprentissage profond.
Segmentation automatique – Méthodes fondées sur un modèle génératif
Dans les méthodes fondées sur un modèle génératif, la segmentation d'une image est modélisée comme un problème d'inférence statistique. Plus précisément, on spécifie un modèle génératif de l'image, dans lequel la segmentation est une variable latente et la segmentation d'une image correspond à l'inférence de cette variable.
Modèles univariés
Pour ce qui est des points, la méthode univariée modélise les intensités d'image de différents segments en tant que sous-populations à partir d'un modèle par mélanges finis (MMF) avec des distributions d'intensité différentes décrivant les processus de génération de données de différents segments. Dans le cadre de cette modélisation, les intensités d'image appartenant à des segments donnés sont tirées de mélanges distincts. Un des cas particuliers de MMF souvent utilisé dans la segmentation d'image est un modèle de mélange gaussien (MMG) dans lequel les distributions de mélange sont gaussiennes.Footnote 2Footnote 3Footnote 4 Au moyen d'un MMG, nous montrerons comment un modèle génératif peut servir à segmenter une image. L'image est représentée par une variable aléatoire qui est un ensemble de variables aléatoires indépendantes avec un support et l'on suppose que est une variable aléatoire avec un support représentant la valeur d'intensité et l'affectation de mélange pour un élément de domaine d'image respectivement. Soit un vecteur de variable aléatoire avec un support comme probabilités de mélange, où la entrée indique la probabilité d'appartenance au segment d'image. En supposant une configuration bayésienne, est une variable latente telle que cet événement indique que appartient au segment d'image et qu'il existe une loi a priori de Dirichlet sur les probabilités de mélange, à savoir La fonction de densité conjointe de probabilité d'affectation de segment et d'intensité à une valeur donnée a la forme suivante :
où et sont des ensembles de paramètres de moyenne et de variance, respectivement, pour chaque mélange et représente des paramètres de parcimonie de Dirichlet.
Modèles multivariés
Contrairement aux méthodes univariées, les méthodes multivariées modélisent la distribution d'intensité sur l'ensemble d'un domaine d'image, tout en tenant compte des dépendances à long terme entre pixels. Les champs aléatoires de Markov (CAM) sont une classe de modèles qui spécifient une distribution dans le domaine de l'image en commençant par discrétiser le domaine de manière à ce que
pour certains and Après cette discrétisation, la segmentation et les intensités sont indexées par les sommets d'un graphe non orienté et par les sommets adjacents correspondant aux pixels adjacents.Footnote 5 Supposons d'abord que les estimations d'intensité moyenne pour chaque classe de segments sont données, puis soit et be the mean and variance intensity estimate for the segment d'image. Nous pouvons alors définir une fonctionnelle qui correspond à la probabilité négative du modèle et qui a la forme suivante :
où est l'ensemble de sommets adjacents à est une fonction indicatrice et est un terme de pénalité qui pénalise les emplacements voisins dans le domaine d'image qui n'ont pas en commun les étiquettes de segment. En pratique sont généralement obtenus par un étiquetage partiel (semi-supervisé) effectué par un spécialiste du domaine. Dans l'équation précédente, la première double sommation pénalise un qui étiquette les pixels dont les intensités s'écartent considérablement de l'intensité moyenne dans cette classe de segments. Pour la classe de segments, la distance par rapport à la moyenne est normalisée par l'écart-type afin que la proximité de la moyenne entre classes de segments soit comparable. La deuxième sommation double favorise un qui donne aux pixels voisins la même étiquette, et est un paramètre qui équilibre les deux sommations doubles.
Inférence et apprentissage
Comme cela a été dit plus haut, dans le contexte des modèles génératifs, la segmentation d'image est un problème statistique dans lequel la segmentation est inférée et les paramètres qui régissent le modèle génératif sont appris. Dans cette sous-section, nous donnons des exemples de problèmes d'inférence qui se posent couramment dans la segmentation d'image.
Maximum de vraisemblance et estimation du maximum a posteriori
Si nous avons accès à une distribution a posteriori ou une vraisemblance soluble, cette inférence est réalisable au moyen d'une estimation par la méthode du maximum de vraisemblance (EMV) ou d'une estimation du maximum a posteriori (MAP) de l'affectation des segments. De façon plus formelle, dans l'hypothèse d'un contexte bayésien, supposons que nous avons accès à une distribution a posteriori soluble et au comportement raisonnable où et sont respectivement le MAP de la segmentation et l'image. L'estimation par le MAP du MAP de la segmentation aurait la forme suivante :
Il n'est pas toujours facile de tirer un échantillon en cas de distribution a posteriori d'une segmentation; cela est particulièrement vrai pour les CAM. Dans ces scénarios, on utilise généralement des méthodes Monte Carlo par chaînes de Markov (MCMC) fondées sur des graphes. Plus précisément, un échantillonnage de Gibbs est généralement utilisé pour les CAMFootnote 6 puisqu'il s'agit d'un cas particulier de champ aléatoire conditionnel (CAC), ce qui permet de relativement facilement spécifier chaque affectation de segment comme une probabilité conditionnelle.
Inférence variationnelle
Plutôt que d'échantillonner une distribution a posteriori insoluble, on peut utiliser une méthode dite d'inférence variationnelle (IV) pour estimer la distribution a posteriori au moyen d'une distribution issue d'une famille de distributions solubles. Cette famille de distributions solubles est celle des distributions variationnelles, d'après le calcul des variations ou calcul variationnel. Après spécification de la famille de distributions, on peut estimer la distribution a posteriori en trouvant la distribution variationnelle qui optimise certaines mesures entre la distribution a posteriori vraie et elle-même. L'indicateur le plus couramment utilisé pour mesurer la similarité entre deux distributions est la divergence Kullback-Leibler (KL), définie comme suit :
où est une densité approximative et est une densité vraie sur le même support. L'inférence de la segmentation latente au moyen de cette approximation de distribution peut être formulée comme un problème d'inférence d'espérance-maximisation bayésienne variationnelle (EMBV).Footnote 7 Une analyse plus approfondie de l'inférence variationnelle peut se trouver dans la section 4 de Variational Inference.Footnote 8
Méthodes d'apprentissage profond
Depuis quelques années, les méthodes d'apprentissage profond (AP) ont été appliquées avec succès à de nombreuses tâches d'apprentissage. Il a été démontré qu'elles surpassent des techniques antérieures d'apprentissage automatique de pointe, en particulier dans le domaine de la vision par ordinateur.Footnote 9 Vaguement inspirées des modèles informatiques d'apprentissage biologique, les méthodes d'AP donnent des modèles informatiques efficaces et extrêmement parallélisables de couches de traitement multiples qui apprennent implicitement des représentations de données.Footnote 10 Les configurations structurelles de ces couches de traitement sont connues sous le nom d'architectures. Certaines des architectures prédominantes dans la vision par ordinateur comprennent les réseaux antagonistes génératifs (ou GAN pour l'anglais)Footnote 11 les réseaux de neurones récurrents (ou RNN en anglais)Footnote 12 et les réseaux neuronaux convolutifs Footnote 13 ces derniers affichant des performances particulièrement bonnes en segmentation d'image. Un réseau neuronal convolutif en 3D appliqué à la segmentation des lésions cérébralesFootnote 14 a permis d'améliorer le modèle précédent pour obtenir les meilleures performances des ensembles de données de référence publics BRATS 2015Footnote 15 et ISLES 2015Footnote 16 (ensembles de données publics utilisés dans les défis sur la segmentation des lésions cérébrales).
Réseaux neuronaux convolutifs
Figure 2 : Processus typique de segmentation par apprentissage profond avec un réseau neuronal convolutif. Source de l'image : Going Deep in Medical Image Analysis: Concepts, Methods, Challenges and Future DirectionsFootnote 17
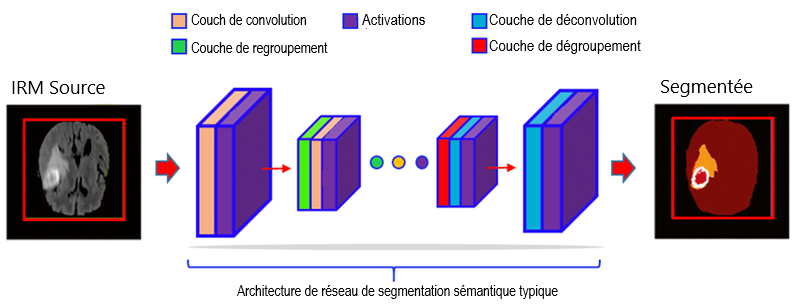
Description - Figure 2 : Processus typique de segmentation par apprentissage profond avec un réseau neuronal convolutif. Source de l'image : Going Deep in Medical Image Analysis: Concepts, Methods, Challenges and Future Directions.
Le processus typique de segmentation par apprentissage profond avec un modèle fondé sur un réseau neuronal convolutif consiste d'abord à comprimer l'image source avec une pile de couches de convolution, d'activation et de regroupement différentes.
À l'heure actuelle, les réseaux neuronaux convolutifs sont considérés comme les réseaux les plus à la pointe de la technologie pour les problèmes de segmentation d'image par AP supervisé.Footnote 18 Leur architecture s'inspire d'un modèle de champ récepteur hiérarchique du cortex visuel et comprend généralement la composition de trois types de couches :
- couches de convolution, où un noyau (filtre) est convolué sur des entrées pour extraire une hiérarchie de caractéristiques;
- couches non linéaires, qui permettent de mapper les entrées aux espaces de caractéristiques;
- couches de regroupement, qui réduisent la résolution spatiale en agrégeant les informations locales.
Chaque couche est constituée d'unités de traitement connectées localement. Ces connexions locales sont appelées champs récepteurs. Les couches sont généralement composées pour former une pyramide multirésolution dans laquelle les couches de niveau supérieur apprennent les caractéristiques de champs récepteurs plus larges. Les paramètres du modèle sont généralement appris au moyen d'une version stochastique de l'algorithme de rétropropagationFootnote 19 qui est une routine d'optimisation par gradient propageant efficacement le gradient du résidu à travers le réseau.
Méthodes d'évaluation
En général, les méthodes d'évaluation de la segmentation supervisée tentent de quantifier le degré de chevauchement entre une segmentation estimée et la segmentation réelle sur le terrain. En utilisant la notation cartographique d'une segmentation de l'expression (2), nous pouvons comprendre de façon équivalente la segmentation comme un ensemble avec l'image de sa carte, c'est-à-dire
Le coefficient de Dice (D) est l'une des méthodes d'évaluation de la segmentation les plus populaires et les plus faciles à comprendre sur le plan conceptuel. Pour deux segmentations et , le coefficient de Dice est calculé comme suit
Le coefficient de Jaccard (J) — une autre méthode d'évaluation de la segmentation — est lié au coefficient de Dice par l'expression suivante
est connu pour produire des valeurs plus grandes pour des volumes plus élevés. Une autre méthode d'évaluation de la segmentation est la distance de Hausdorff moyenne, qui est particulièrement recommandée pour les tâches de segmentation présentant des limites complexes et de petits segments minces. Comparativement au coefficient de Dice, la distance de Hausdorff moyenne a l'avantage de tenir compte de la localisation quand on considère les performances de la segmentationFootnote 20 Pour deux segmentations et , qui sont des sous-ensembles non vides d'un espace métrique , la distance de Hausdorff moyenne est calculée comme suit :
Pour connaître d'autres méthodes d'évaluation, se reporter à Metrics for evaluating 3D medical image segmentationFootnote 22
Conclusion
En conclusion, la segmentation d'image est une technique cruciale dans le traitement d'image en général et en imagerie médicale en particulier. Ce processus est une partie essentielle d'un pipeline de traitement d'image qui nécessite des analyses d'images en aval, dans lesquelles sont identifiées les sous-structures sémantiques d'une image. L'apprentissage automatique nous permet d'automatiser cette procédure et de conserver la qualité d'exécution d'une annotation par un spécialiste, pour un coût nettement moindre.

Si vous avez des questions à propos de mon article ou si vous souhaitez en discuter davantage, je vous invite à une Rencontre avec le scientifique des données, un événement au cours duquel les auteurs rencontrent les lecteurs, présentent leur sujet et discutent de leurs résultats.
Jeudi, le 17 novembre
14 h 00 à 15 h 00 HNE
MS Teams – le lien sera fourni aux participants par courriel
Inscrivez-vous à la présentation Rencontre avec le scientifique des données. À bientôt !
- Date de modification :